Zobacz slidy
Treść prezentacji
Slide 1
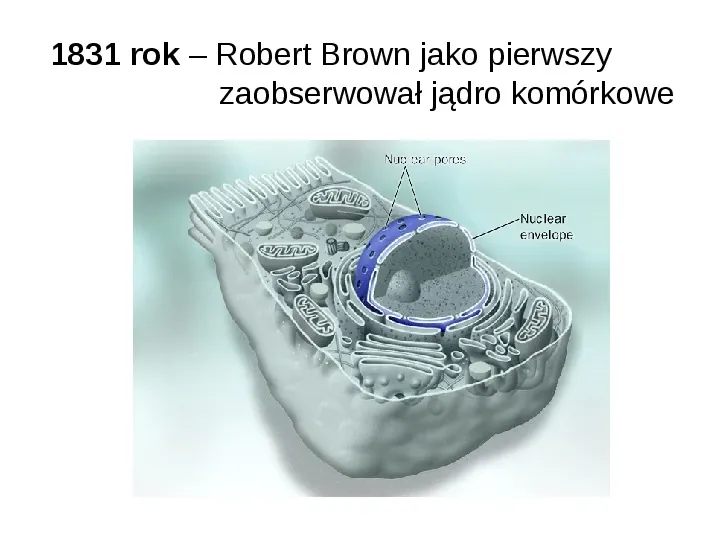
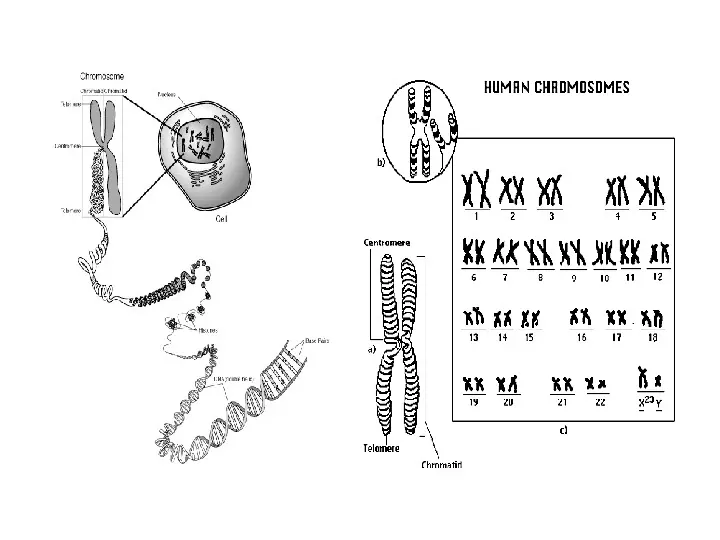
1831 rok Robert Brown jako pierwszy zaobserwował jądro komórkowe
Slide 2
Kształt jądra zależy od kształtu komórki, wieku i sprawności czynnościowej Jest zazwyczaj kuliste w komórkach foremnych (kulistych, 6-cio bocznych lub wielobocznych) a elipsoidalne, gdy ma ona postać walca, graniastosłupa lub wrzeciona. W wielu k. człowieka są rogalikowate czy segmentowane - granulocyty, we włóknach mięśniowych wydłużone. J. komórek dzielących się lub bezpośrednio po podziale są na ogół kuliste. K. dojrzałe, zróżnicowane i starsze mają bardziej zniekształcone jądra. kuliste w komórkach foremnych, elipsoidalne w komórkach o kształcie walca, graniastosłupa lub wrzeciona
Slide 3
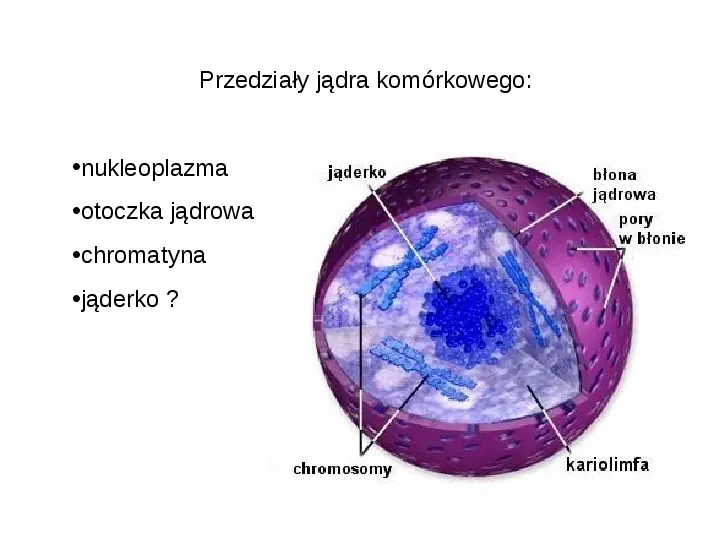
Przedziały jądra komórkowego: nukleoplazma otoczka jądrowa chromatyna jąderko ?
Slide 4
Jąderko Podstawową funkcją jąderka jest synteza rybosomowych prekursorów RNA (pre-RNA), montaż i dojrzewanie cząsteczek prerybosomowych, które są prekursorami dojrzałych rybosomów cytoplazmatycznych. Reaguje błyskawicznie na zapotrzebowanie komórki na te miniaturowe wytwórnie białek RYBOSOMY struktury o średnicy 20 nm, złożone zawsze z 2 podjednostek: dużej i małej obie podjednostki zbudowane są z rRNA i białek występują w każdej komórce (oprócz dojrzałych erytrocytów)
Slide 5
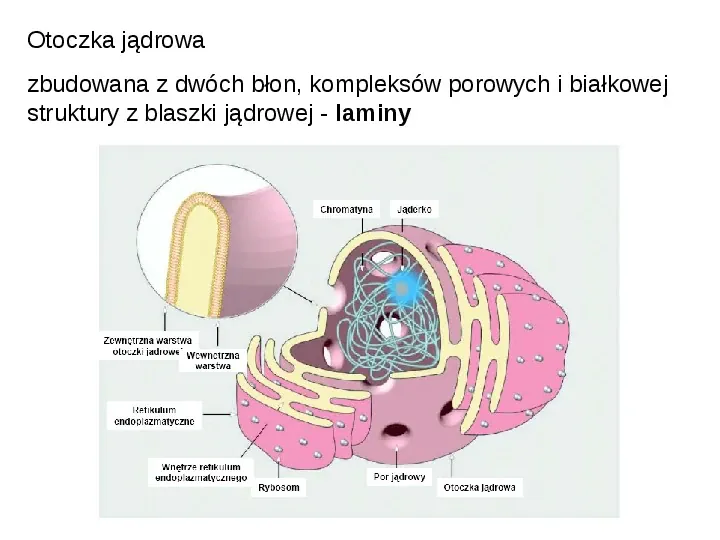
Otoczka jądrowa zbudowana z dwóch błon, kompleksów porowych i białkowej struktury z blaszki jądrowej - laminy
Slide 6
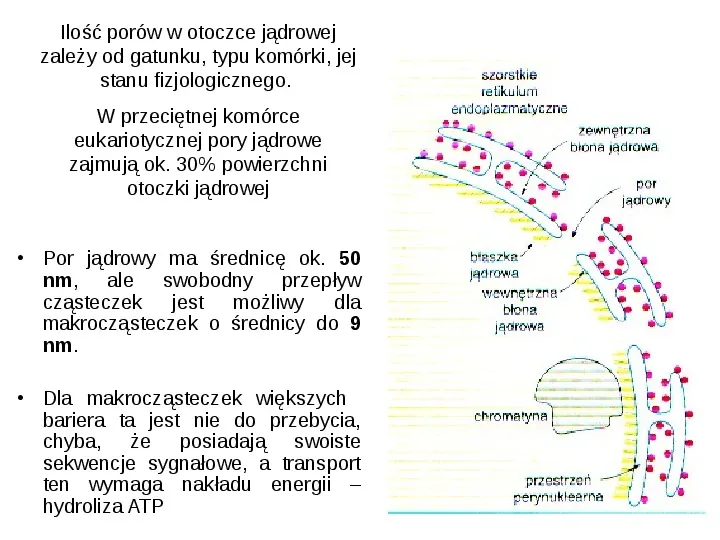
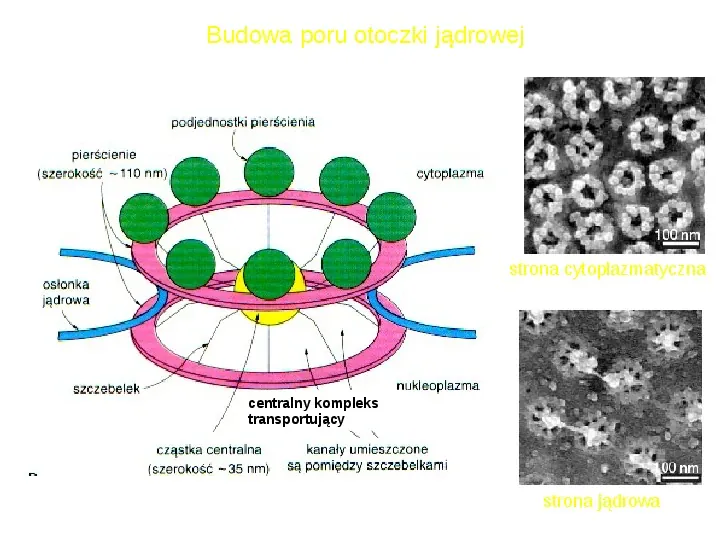
Ilość porów w otoczce jądrowej zależy od gatunku, typu komórki, jej stanu fizjologicznego. W przeciętnej komórce eukariotycznej pory jądrowe zajmują ok. 30 powierzchni otoczki jądrowej Por jądrowy ma średnicę ok. 50 nm, ale swobodny przepływ cząsteczek jest możliwy dla makrocząsteczek o średnicy do 9 nm. Dla makrocząsteczek większych bariera ta jest nie do przebycia, chyba, że posiadają swoiste sekwencje sygnałowe, a transport ten wymaga nakładu energii hydroliza ATP
Slide 7
Budowa poru otoczki jądrowej strona cytoplazmatyczna centralny kompleks transportujący strona jądrowa
Slide 8
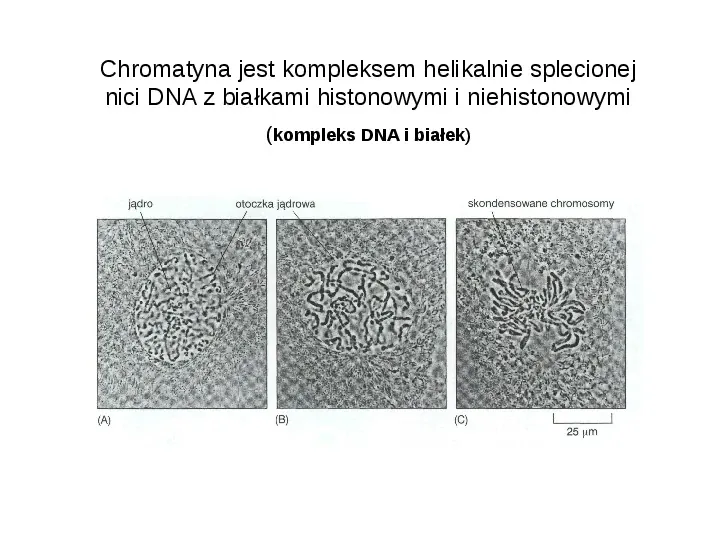
Chromatyna jest kompleksem helikalnie splecionej nici DNA z białkami histonowymi i niehistonowymi (kompleks DNA i białek)
Slide 9
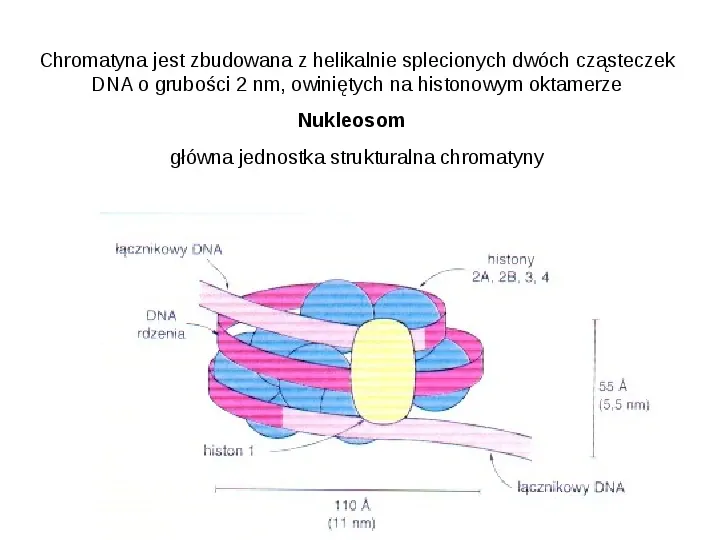
Chromatyna jest zbudowana z helikalnie splecionych dwóch cząsteczek DNA o grubości 2 nm, owiniętych na histonowym oktamerze Nukleosom główna jednostka strukturalna chromatyny
Slide 10
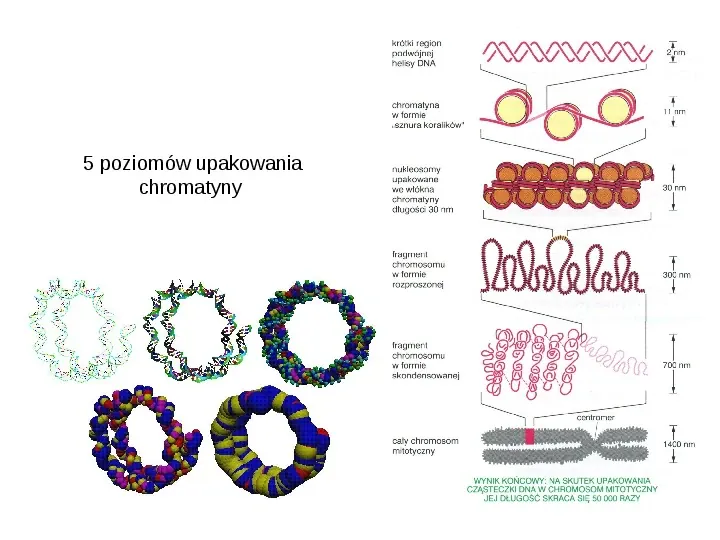
5 poziomów upakowania chromatyny
Slide 12
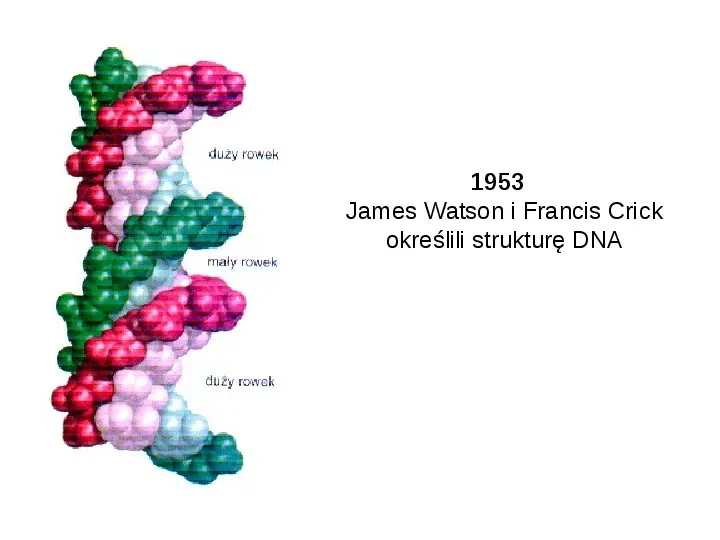
1953 James Watson i Francis Crick określili strukturę DNA
Slide 13
Odkrycie struktury DNA było przełomowym momentem w biologii po nim nastąpiło zrozumienie wszystkich podstawowych procesów życiowych procesy bezpośrednio związane z DNA zostały przewidziane przez model albo dzięki niemu szybko odkryte w fizjologii, biochemii i biologii rozwoju nastąpiło znaczące przyśpieszenie
Slide 14
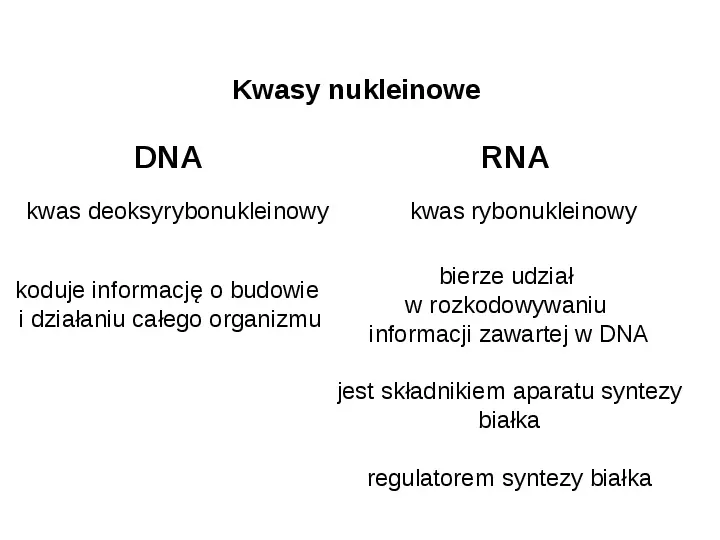
Kwasy nukleinowe DNA kwas deoksyrybonukleinowy koduje informację o budowie i działaniu całego organizmu RNA kwas rybonukleinowy bierze udział w rozkodowywaniu informacji zawartej w DNA jest składnikiem aparatu syntezy białka regulatorem syntezy białka
Slide 15
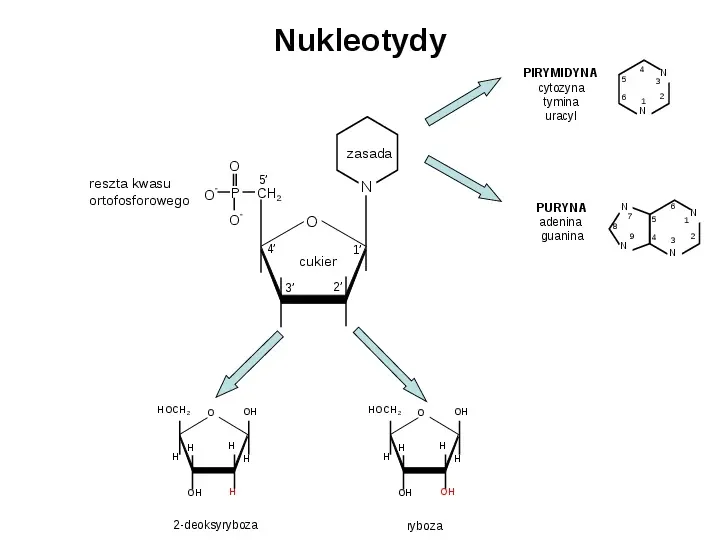
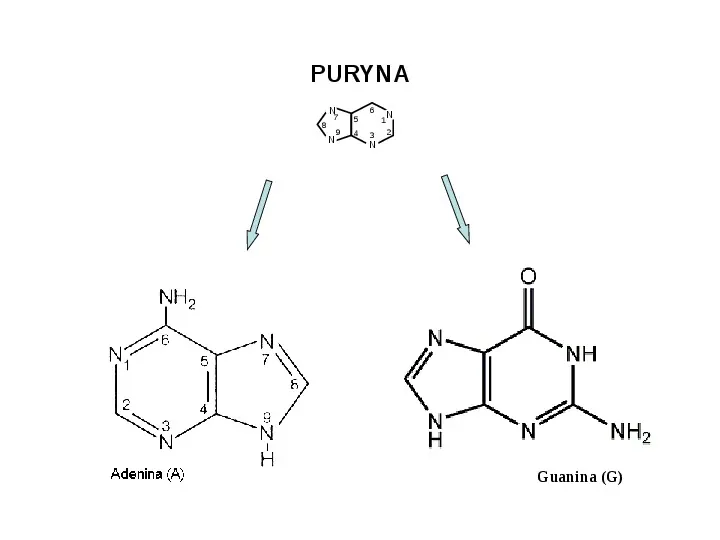
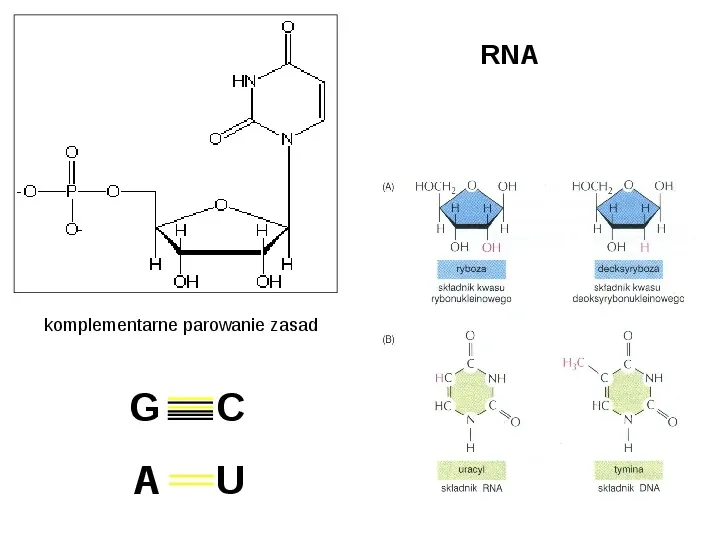
Nukleotydy 4 PIRYMIDYNA cytozyna tymina uracyl O - 5 P O - H H OH cukier 1 2 HOCH2 H H OH PURYNA adenina guanina O 3 O 6 2 1 N N CH2 4 HOCH2 N 3 zasada O reszta kwasu ortofosforowego 5 H 2-deoksyryboza H OH O H H H OH OH ryboza N 6 7 5 9 4 8 N 1 3 N N 2
Slide 16
PURYNA N 8 6 7 5 9 4 N 1 3 N 2 N Guanina (G)
Slide 17
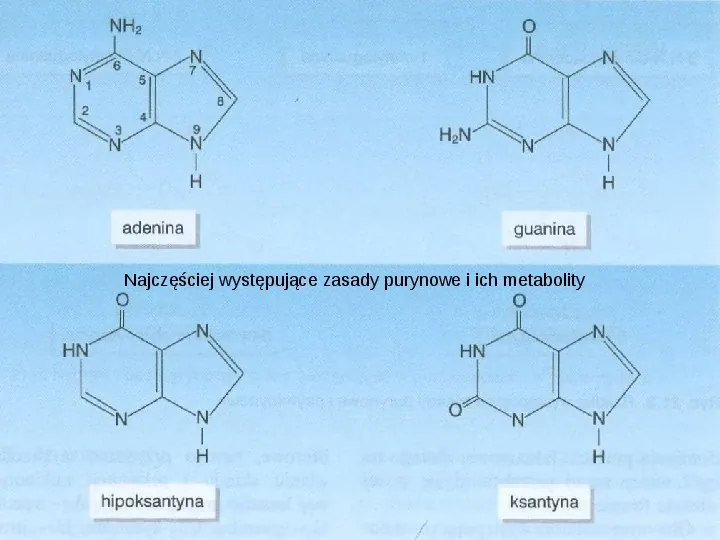
Najczęściej występujące zasady purynowe i ich metabolity
Slide 18
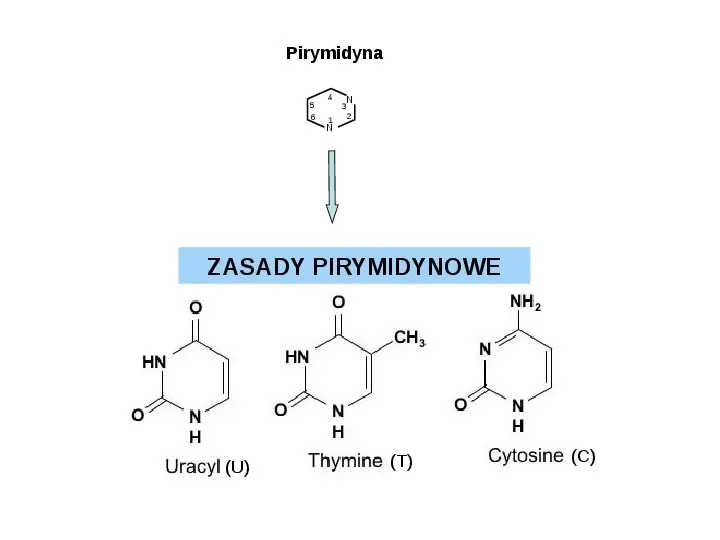
Pirymidyna 5 6 4 1 N 3 2 N ZASADY PIRYMIDYNOWE (U) (T) (C)
Slide 19
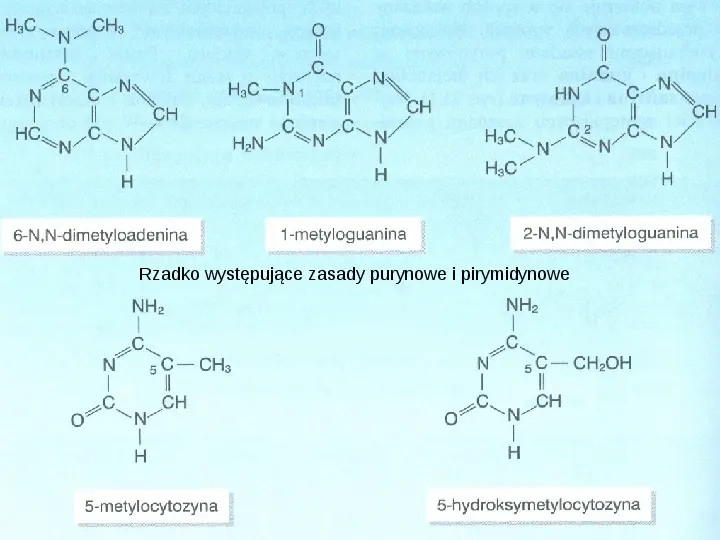
Rzadko występujące zasady purynowe i pirymidynowe
Slide 20
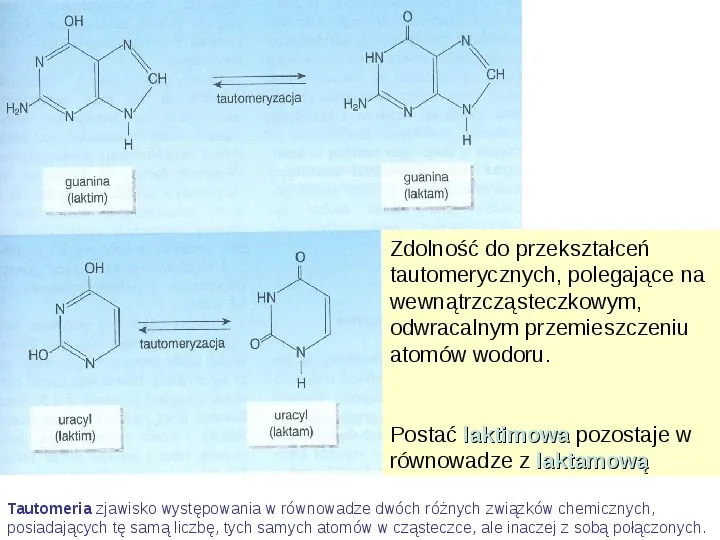
Zdolność do przekształceń tautomerycznych, polegające na wewnątrzcząsteczkowym, odwracalnym przemieszczeniu atomów wodoru. Postać laktimowa pozostaje w równowadze z laktamową Tautomeria zjawisko występowania w równowadze dwóch różnych związków chemicznych, posiadających tę samą liczbę, tych samych atomów w cząsteczce, ale inaczej z sobą połączonych.
Slide 21
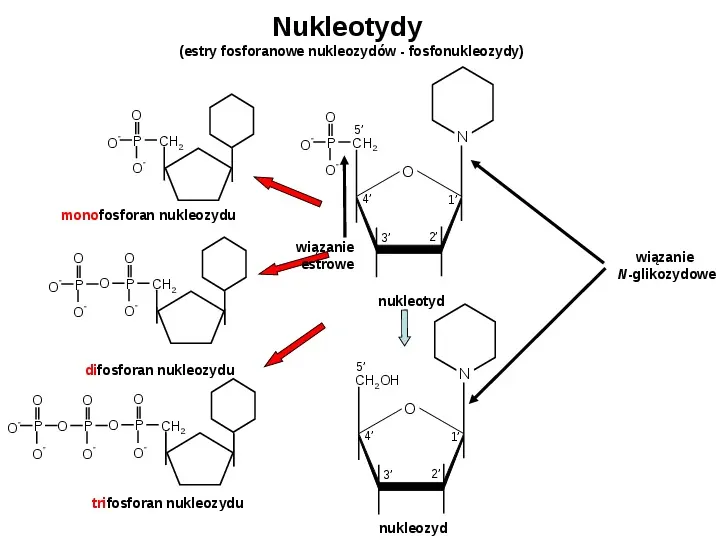
Nukleotydy (estry fosforanowe nukleozydów - fosfonukleozydy) O O - O P O CH2 - O - P O 5 N CH2 - O 4 1 monofosforan nukleozydu O O O - O P O - P O O O - O - O P O nukleotyd - 5 - O O P O N CH2OH O O P wiązanie N-glikozydowe CH2 difosforan nukleozydu 2 3 wiązanie estrowe CH2 - 4 1 3 2 trifosforan nukleozydu nukleozyd
Slide 22
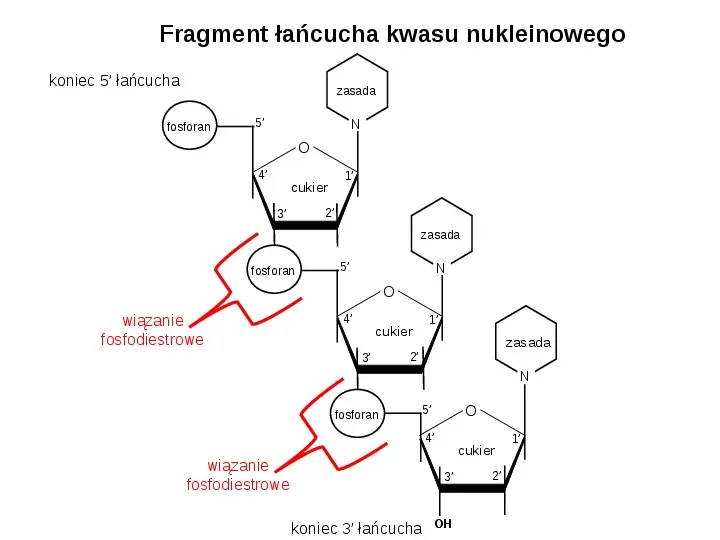
Fragment łańcucha kwasu nukleinowego koniec 5 łańcucha zasada fosforan N 5 O 4 cukier 1 2 3 zasada fosforan N 5 O wiązanie fosfodiestrowe 4 1 cukier 3 zasada 2 N fosforan O 5 4 wiązanie fosfodiestrowe cukier 3 koniec 3 łańcucha OH 2 1
Slide 23
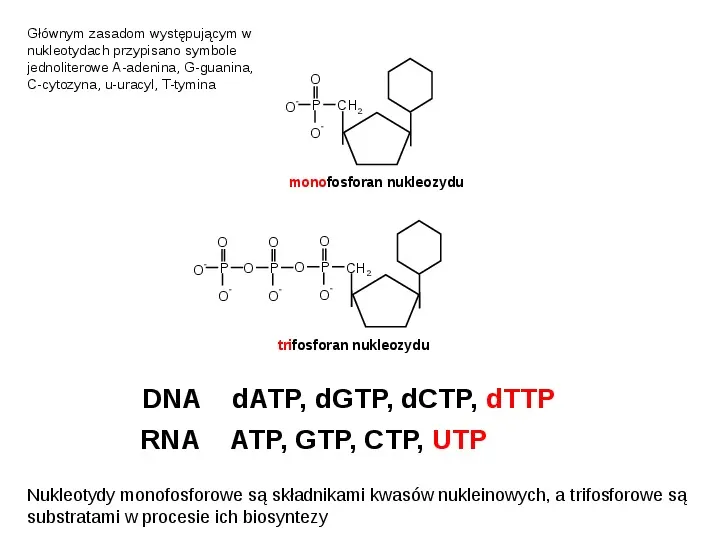
Głównym zasadom występującym w nukleotydach przypisano symbole jednoliterowe A-adenina, G-guanina, C-cytozyna, u-uracyl, T-tymina O O - P O CH2 - monofosforan nukleozydu O O - P O O O - O P O - O P O CH2 - trifosforan nukleozydu DNA dATP, dGTP, dCTP, dTTP RNA ATP, GTP, CTP, UTP Nukleotydy monofosforowe są składnikami kwasów nukleinowych, a trifosforowe są substratami w procesie ich biosyntezy
Slide 25
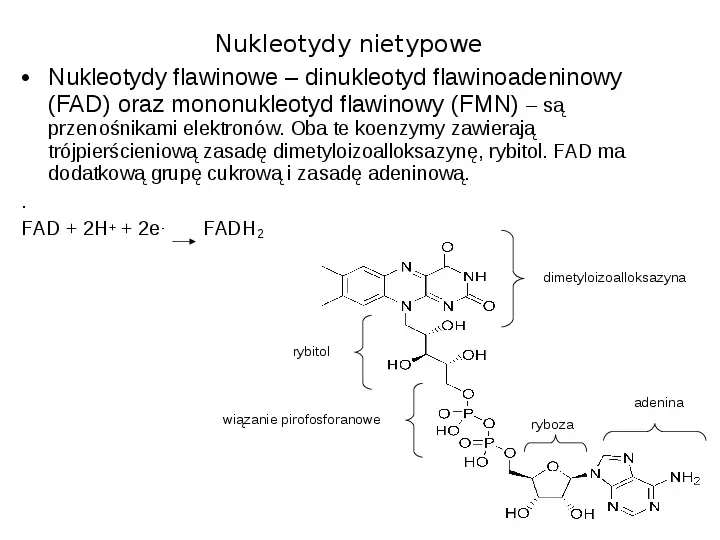
Nukleotydy nietypowe Nukleotydy flawinowe dinukleotyd flawinoadeninowy (FAD) oraz mononukleotyd flawinowy (FMN) są przenośnikami elektronów. Oba te koenzymy zawierają trójpierścieniową zasadę dimetyloizoalloksazynę, rybitol. FAD ma dodatkową grupę cukrową i zasadę adeninową. . FAD 2H 2e- FADH2 dimetyloizoalloksazyna rybitol adenina wiązanie pirofosforanowe ryboza
Slide 26
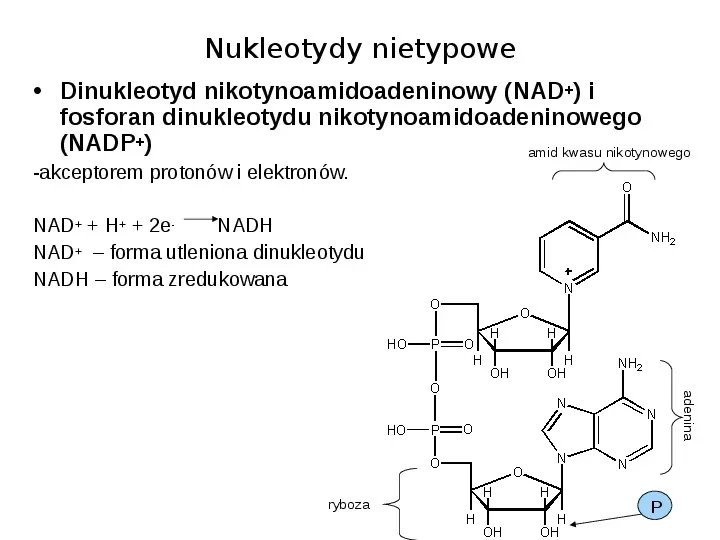
Nukleotydy nietypowe Dinukleotyd nikotynoamidoadeninowy (NAD) i fosforan dinukleotydu nikotynoamidoadeninowego (NADP) amid kwasu nikotynowego -akceptorem protonów i elektronów. NAD H 2eNADH NAD forma utleniona dinukleotydu NADH forma zredukowana adenina ryboza P
Slide 27
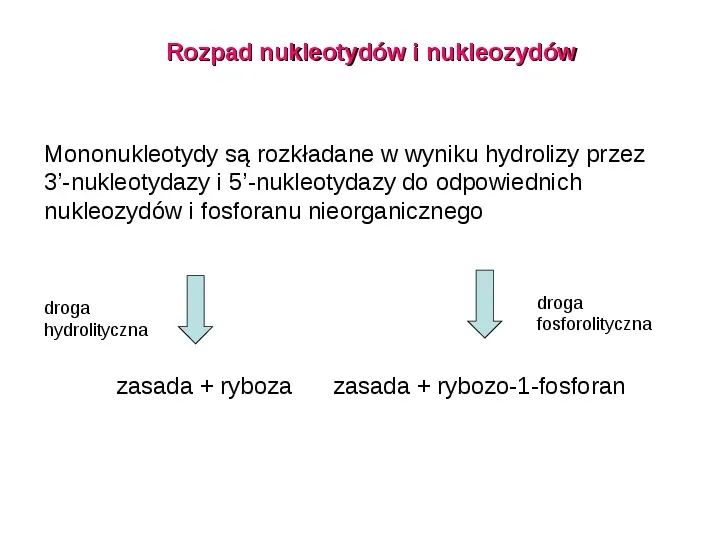
Rozpad nukleotydów i nukleozydów Mononukleotydy są rozkładane w wyniku hydrolizy przez 3-nukleotydazy i 5-nukleotydazy do odpowiednich nukleozydów i fosforanu nieorganicznego droga hydrolityczna zasada ryboza droga fosforolityczna zasada rybozo-1-fosforan
Slide 28
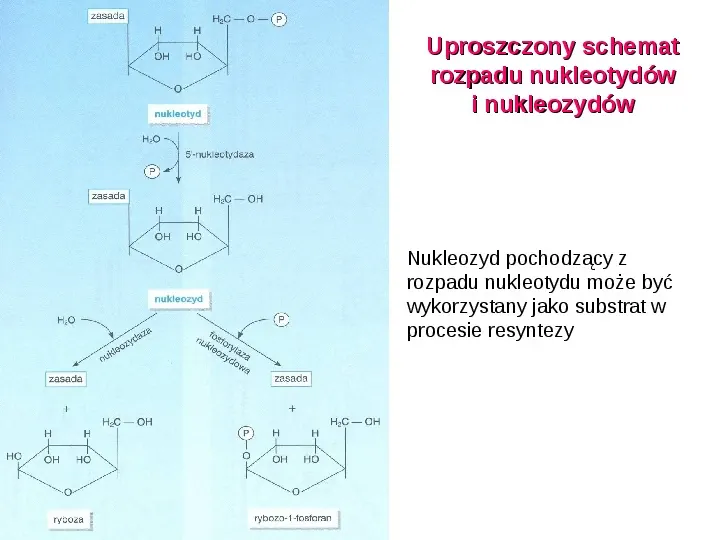
Uproszczony schemat rozpadu nukleotydów i nukleozydów Nukleozyd pochodzący z rozpadu nukleotydu może być wykorzystany jako substrat w procesie resyntezy
Slide 29
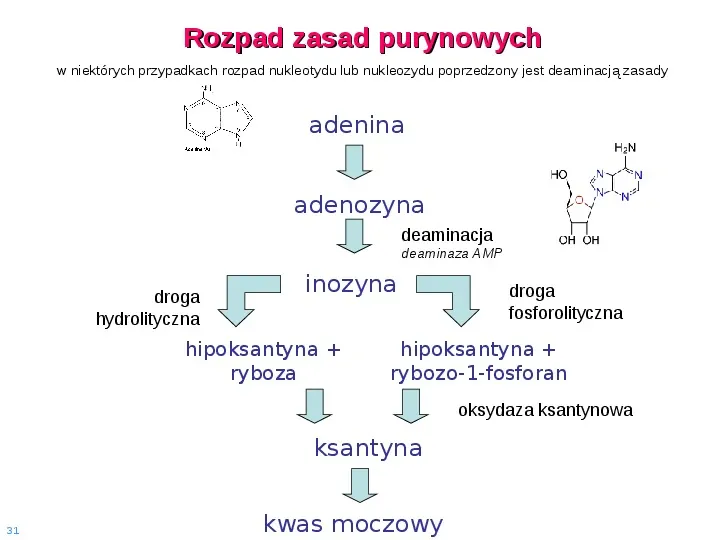
Rozpad zasad purynowych w niektórych przypadkach rozpad nukleotydu lub nukleozydu poprzedzony jest deaminacją zasady adenina adenozyna deaminacja deaminaza AMP droga hydrolityczna inozyna hipoksantyna ryboza droga fosforolityczna hipoksantyna rybozo-1-fosforan oksydaza ksantynowa ksantyna 31 kwas moczowy
Slide 30
Rozpad zasad purynowych guanina deaminaza guaninowa ksantyna oksydaza ksantynowa kwas moczowy Kwas moczowy jest końcowym produktem metabolizmu puryn u człowieka. Pełni rolę antyoksydanta unieczynnia reaktywne formy tlenu
Slide 31
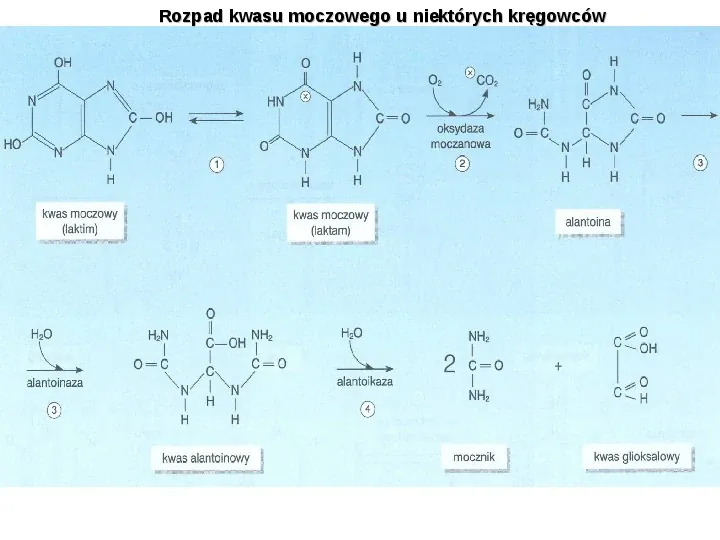
Rozpad kwasu moczowego u niektórych kręgowców
Slide 32
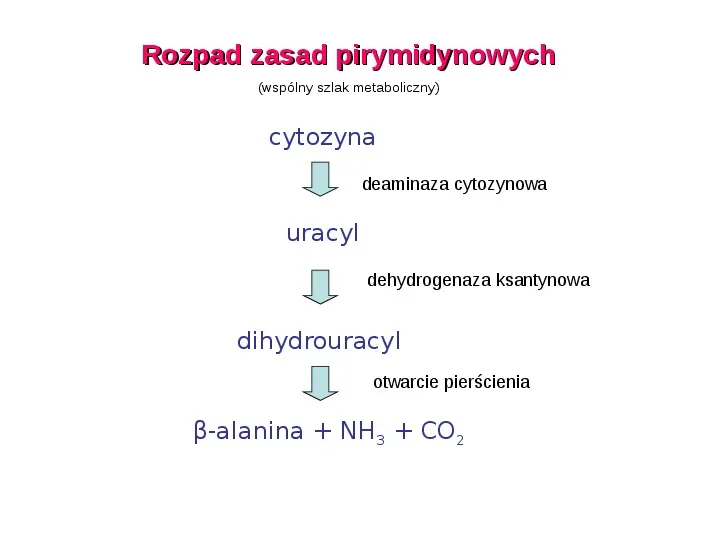
Rozpad zasad pirymidynowych (wspólny szlak metaboliczny) cytozyna deaminaza cytozynowa uracyl dehydrogenaza ksantynowa dihydrouracyl otwarcie pierścienia β-alanina NH3 CO2
Slide 33
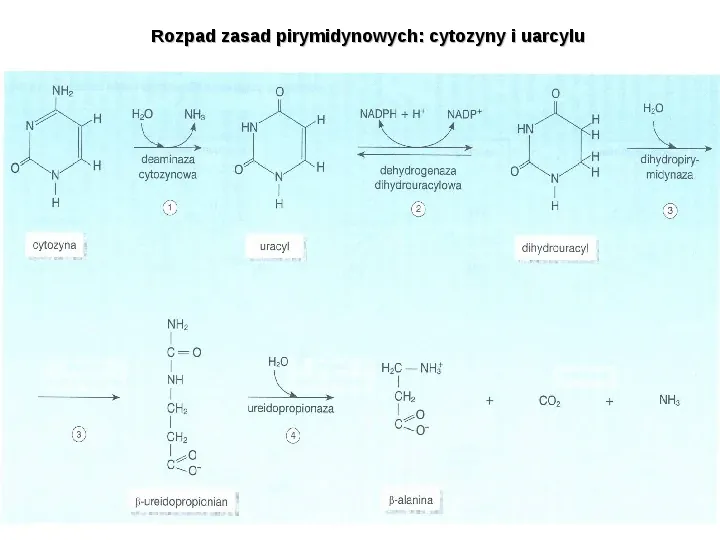
Rozpad zasad pirymidynowych: cytozyny i uarcylu
Slide 34
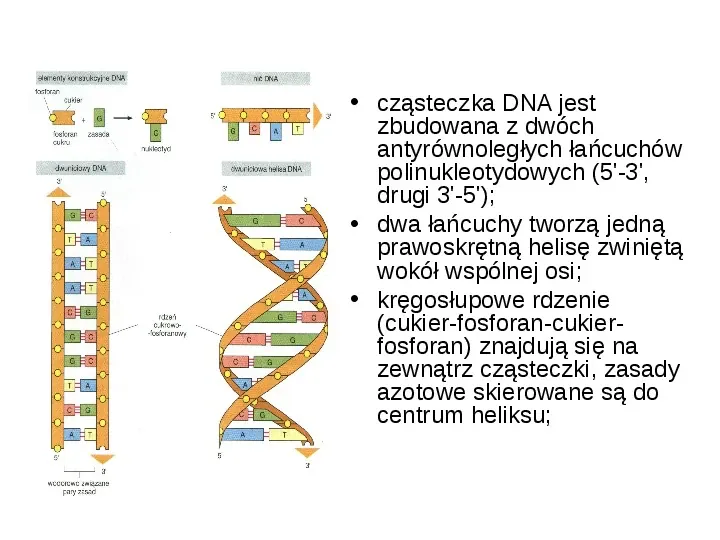
cząsteczka DNA jest zbudowana z dwóch antyrównoległych łańcuchów polinukleotydowych (5-3, drugi 3-5); dwa łańcuchy tworzą jedną prawoskrętną helisę zwiniętą wokół wspólnej osi; kręgosłupowe rdzenie (cukier-fosforan-cukierfosforan) znajdują się na zewnątrz cząsteczki, zasady azotowe skierowane są do centrum heliksu;
Slide 35
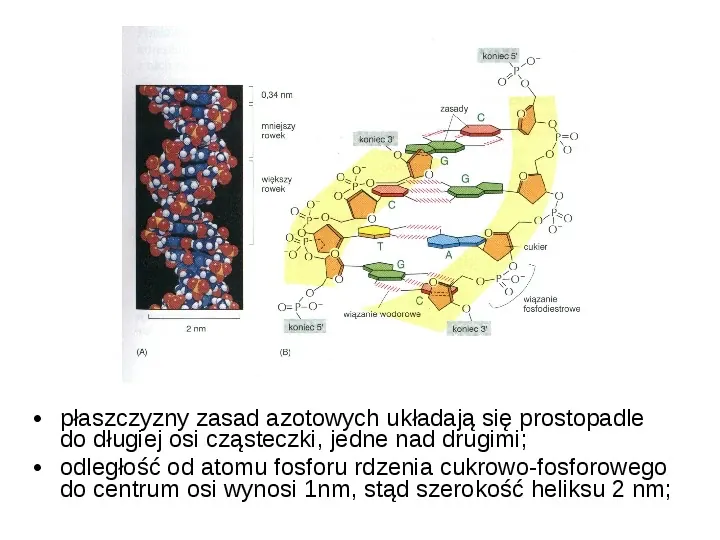
płaszczyzny zasad azotowych układają się prostopadle do długiej osi cząsteczki, jedne nad drugimi; odległość od atomu fosforu rdzenia cukrowo-fosforowego do centrum osi wynosi 1nm, stąd szerokość heliksu 2 nm;
Slide 36
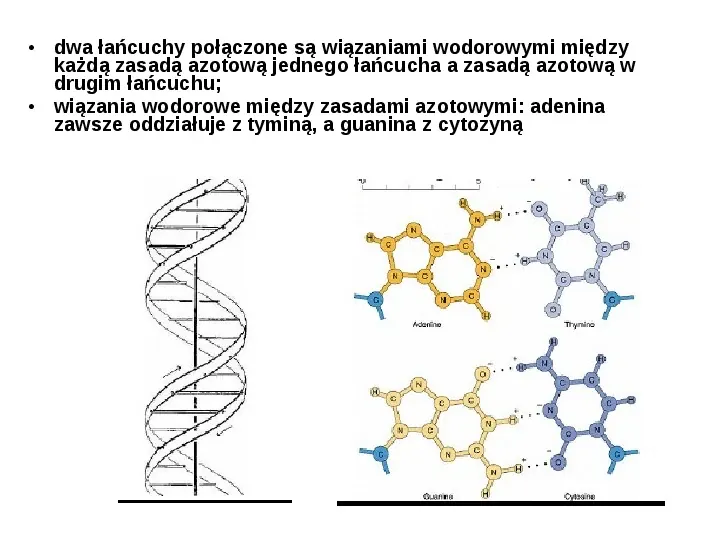
dwa łańcuchy połączone są wiązaniami wodorowymi między każdą zasadą azotową jednego łańcucha a zasadą azotową w drugim łańcuchu; wiązania wodorowe między zasadami azotowymi: adenina zawsze oddziałuje z tyminą, a guanina z cytozyną
Slide 37
podwójna helisa zawiera około 10 par nukleotydów na jeden skręt; sekwencja nukleotydów w jednym z obu łańcuchów DNA może być dowolna, ale kolejność zasad jednego łańcucha determinuje sekwencję zasad drugiego (komplementarność zasad)
Slide 38
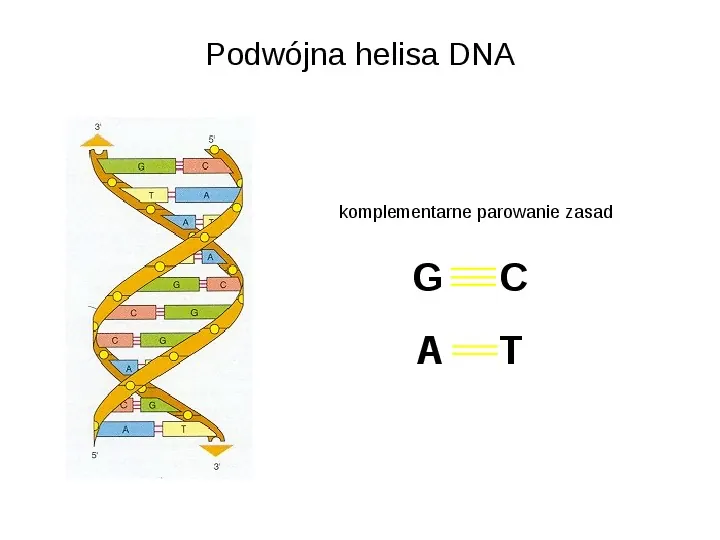
Podwójna helisa DNA komplementarne parowanie zasad G C A T
Slide 39
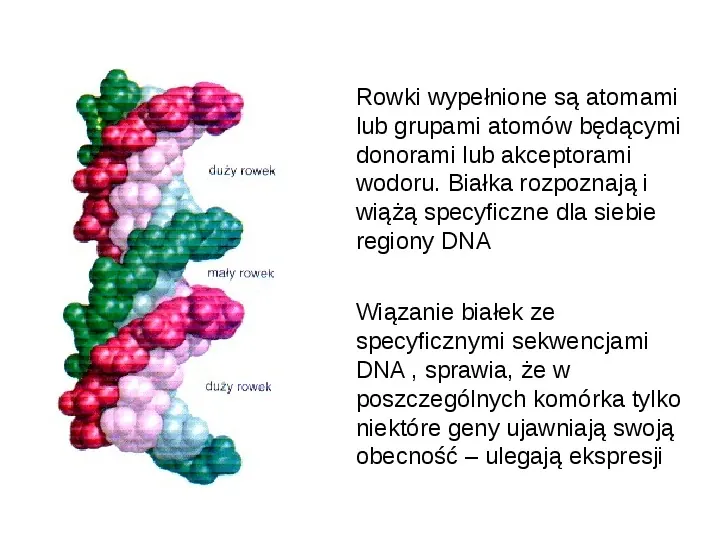
Rowki wypełnione są atomami lub grupami atomów będącymi donorami lub akceptorami wodoru. Białka rozpoznają i wiążą specyficzne dla siebie regiony DNA Wiązanie białek ze specyficznymi sekwencjami DNA , sprawia, że w poszczególnych komórka tylko niektóre geny ujawniają swoją obecność ulegają ekspresji
Slide 40
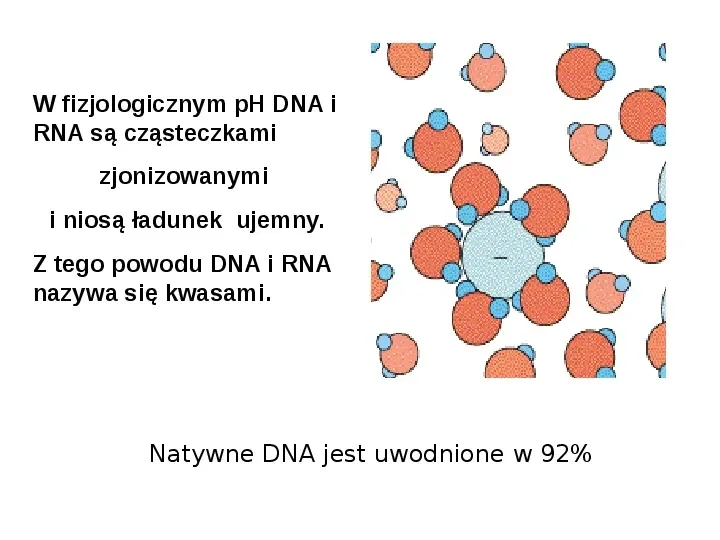
W fizjologicznym pH DNA i RNA są cząsteczkami zjonizowanymi i niosą ładunek ujemny. Z tego powodu DNA i RNA nazywa się kwasami. Natywne DNA jest uwodnione w 92
Slide 41
Struktura przestrzenna DNA obejmuje 4 poziomy: pierwszorzędowe DNA kolejność nukleotydów (sekwencja) w łańcuchu polinukleotydowym, drugorzędowe DNA dwa heliksowo zwinięte łańcuchy, trzeciorzędowe DNA specyficzna konfiguracja w przestrzeni wielkocząsteczkowego podwójnego heliksu DNA, czwartorzędowe DNA być może oddziaływanie wzajemne cząsteczek DNA mających swoje określone struktury I, II, IIIrzędowe.
Slide 42
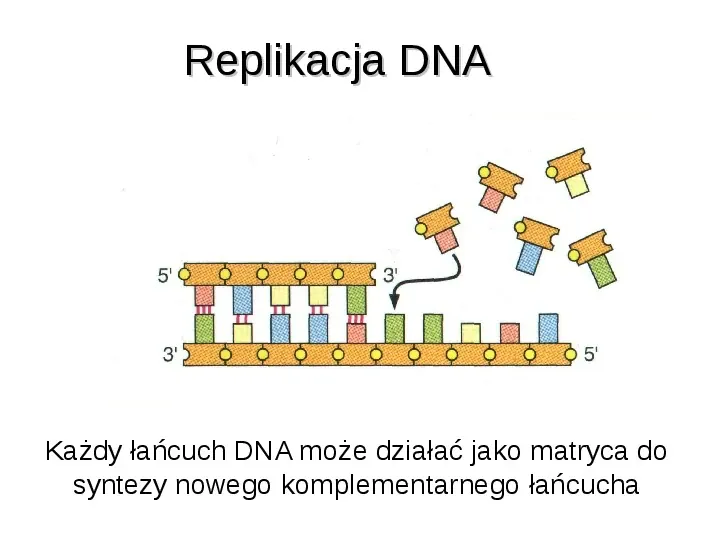
Replikacja DNA Każdy łańcuch DNA może działać jako matryca do syntezy nowego komplementarnego łańcucha
Slide 43
Replikacja DNA synteza dwóch komplementarnych dwuniciowych helis z jednej wyjściowej
Slide 44
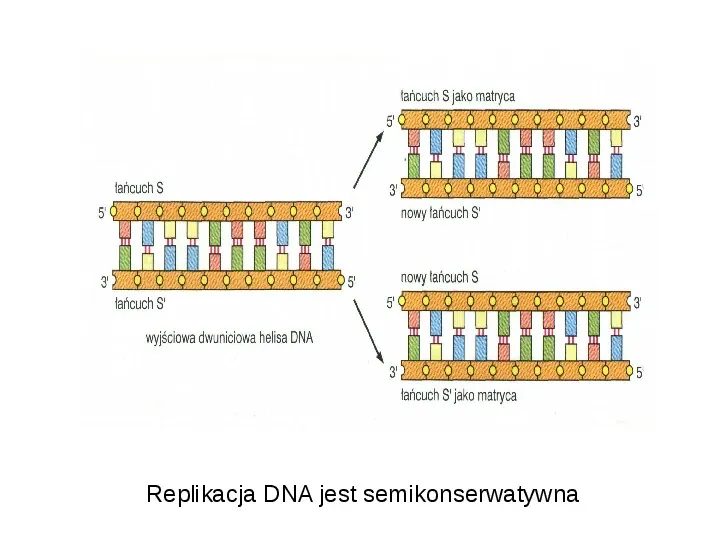
Replikacja DNA jest semikonserwatywna
Slide 45
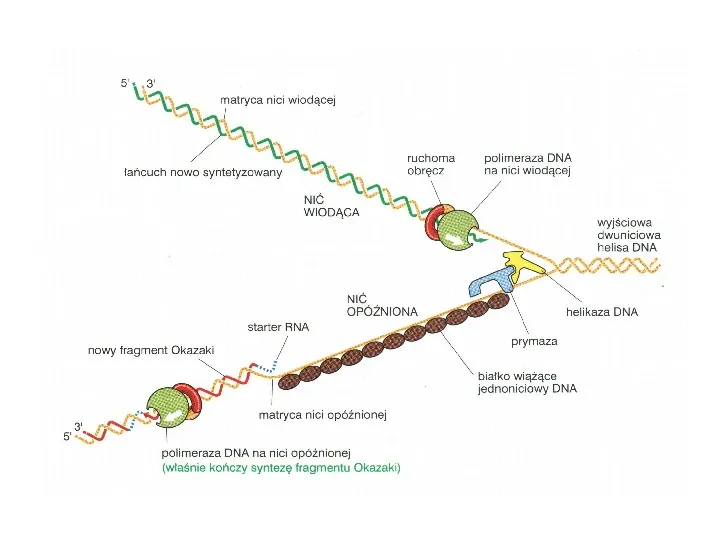
Replikacja DNA jest wieloetapowym procesem enzymatycznym, który zapewnia wierne powielenie jego cząsteczki macierzystej zanim nastąpi podział komórki Zespół białek dokonujących replikacji nazywamy aparatem replikacyjnym Miejsce ori miejsce inicjacji procesu replikacji związanie specyficznych białek inicjujących z DNA, rozpoznanie określonych odcinków DNA zerwanie wiązań wodorowych między zasadami
Slide 46
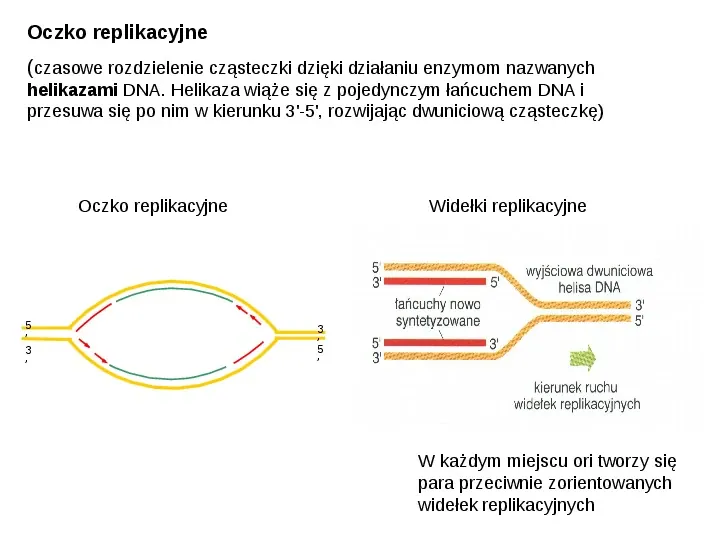
Oczko replikacyjne (czasowe rozdzielenie cząsteczki dzięki działaniu enzymom nazwanych helikazami DNA. Helikaza wiąże się z pojedynczym łańcuchem DNA i przesuwa się po nim w kierunku 3-5, rozwijając dwuniciową cząsteczkę) Oczko replikacyjne 5 3 Widełki replikacyjne 3 5 W każdym miejscu ori tworzy się para przeciwnie zorientowanych widełek replikacyjnych
Slide 47
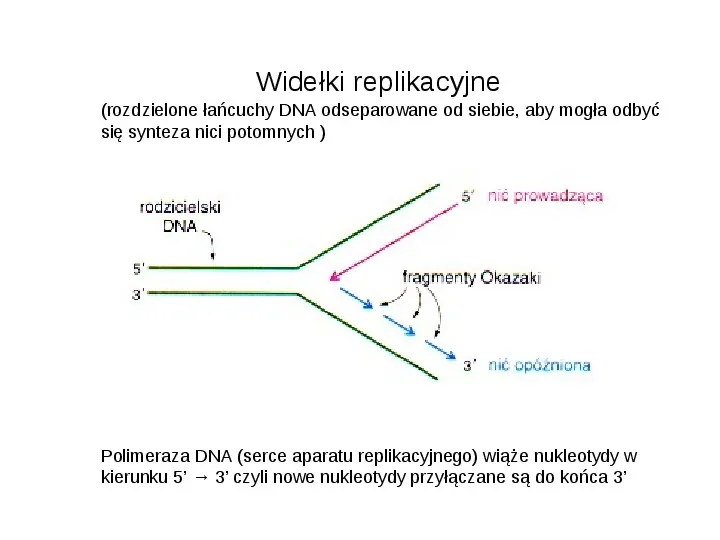
Widełki replikacyjne (rozdzielone łańcuchy DNA odseparowane od siebie, aby mogła odbyć się synteza nici potomnych ) Polimeraza DNA (serce aparatu replikacyjnego) wiąże nukleotydy w kierunku 5 3 czyli nowe nukleotydy przyłączane są do końca 3
Slide 48
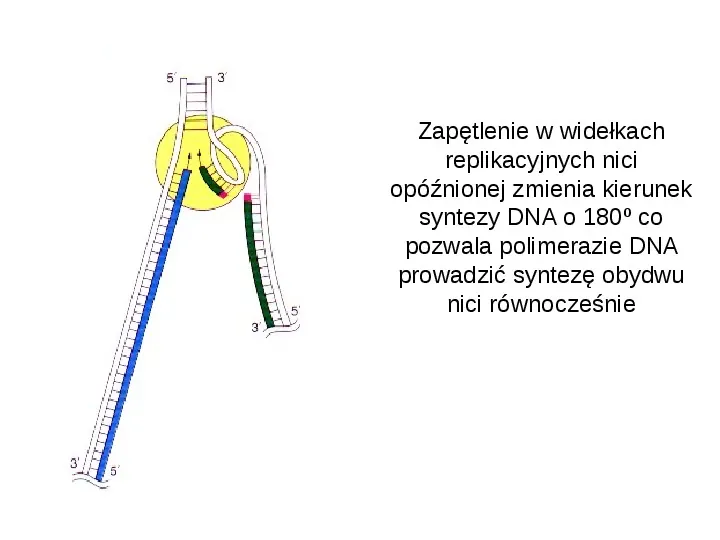
Zapętlenie w widełkach replikacyjnych nici opóźnionej zmienia kierunek syntezy DNA o 180º co pozwala polimerazie DNA prowadzić syntezę obydwu nici równocześnie
Slide 49
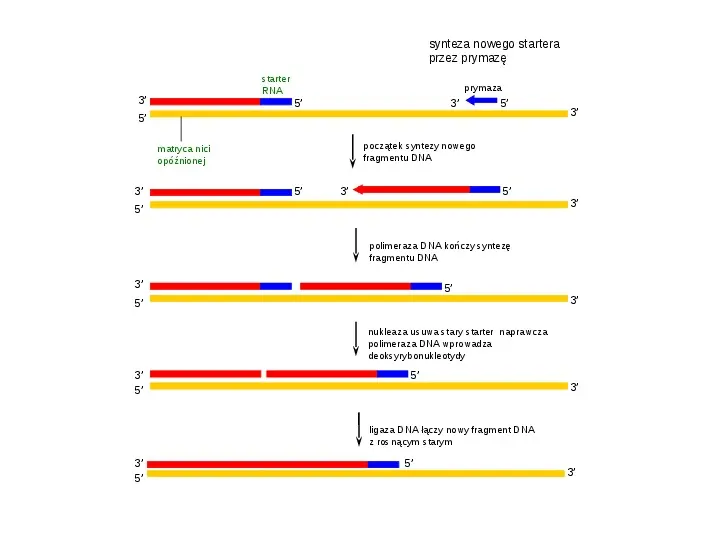
synteza nowego startera przez prymazę starter RNA 3 prymaza 5 3 5 5 początek syntezy nowego fragmentu DNA matryca nici opóźnionej 3 3 5 3 5 5 3 polimeraza DNA kończy syntezę fragmentu DNA 3 5 5 3 nukleaza usuwa stary starter naprawcza polimeraza DNA wprowadza deoksyrybonukleotydy 3 5 5 3 ligaza DNA łączy nowy fragment DNA z rosnącym starym 3 5 5 3
Slide 51
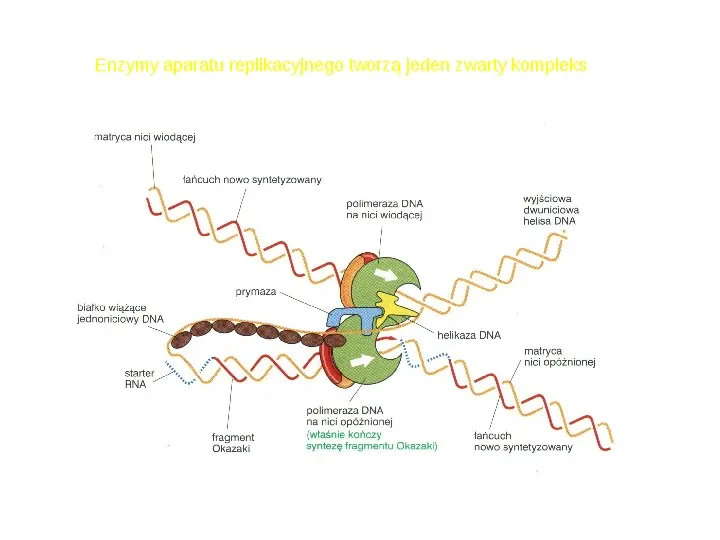
Enzymy aparatu replikacyjnego tworzą jeden zwarty kompleks
Slide 52
Polimeraza DNA jest bardzo dokładna popełnia tylko jeden błąd na 107 poprawnie wprowadzonych nukleotydów. Polimeraza może korygować swoje pomyłki. Ta korekta błędów nazywana jest redagowaniem. Zanim polimeraza dołączy nowy nukleotyd do rosnącego łańcucha, sprawdza czy wcześniej wbudowany nukleotyd utworzył poprawną parę z zasadą matrycy. Polimeraza DNA ma aktywność plimerazową w kierunku 5-3 i endonukleazową w kierunku 3-5.
Slide 53
RNA komplementarne parowanie zasad G C A U
Slide 54
Zazwyczaj RNA występuje w postaci jednoniciowego, silnie pofałdowanego polinukleotydu zawierającego odcinki dwuniciowe i tworzącego skomplikowane struktury przestrzenne. W przeciwieństwie do DNA stosunek zasad azotowych purynowych do pirymidynowych w cząsteczce jednoniciowego RNA nie jest zachowany. Rodzaje RNA Informacyjny kwas rybonukleinowy: mRNA (ok. 5 wszystkich RNA) jest przenośnikiem informacji z jądra do cytoplazmy pełni funkcję matrycy w procesie biosyntezy białek Rybosomowe RNA: rRNA (ok. 80 wszystkich RNA), stanowi rdzeń rybosomów, na którym mRNA ulega translacji do białek Transferowy (transportujący) RNA: tRNA działa jako cząsteczki adaptorowe, które dokonują selekcji aminokwasów i wprowadzają je do białek (związany jest z enzymem - syntetazą aminoacylo-tRNA)
Slide 55
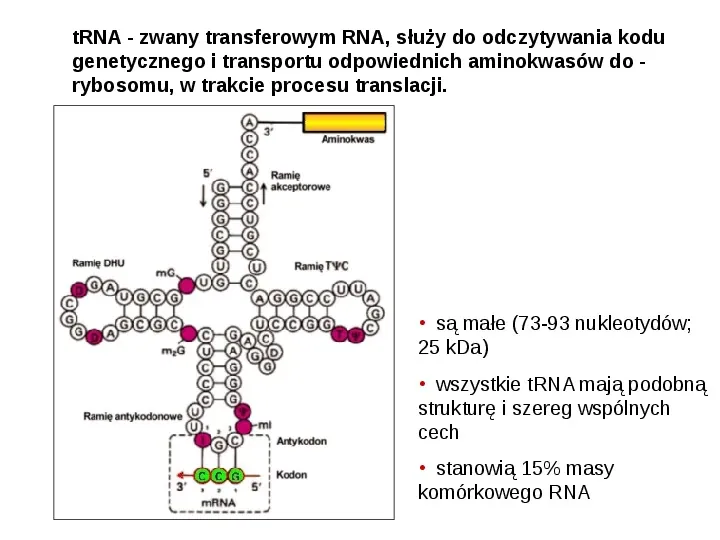
tRNA - zwany transferowym RNA, służy do odczytywania kodu genetycznego i transportu odpowiednich aminokwasów do rybosomu, w trakcie procesu translacji. są małe (73-93 nukleotydów; 25 kDa) wszystkie tRNA mają podobną strukturę i szereg wspólnych cech stanowią 15 masy komórkowego RNA
Slide 56
Budowa cząsteczek tRNA cechy wspólne: koniec 5 jest fosforylowany. Końcowym nukleotydem jest zwykle nukleotyd guanozynowy na końcu 3 - sekwencja CCA Grupa OH przy węglu 3 rybozy końcowego AMP wiąże aminokwas wiązaniem estrowym. Powstaje aminoacylo-tRNA
Slide 57
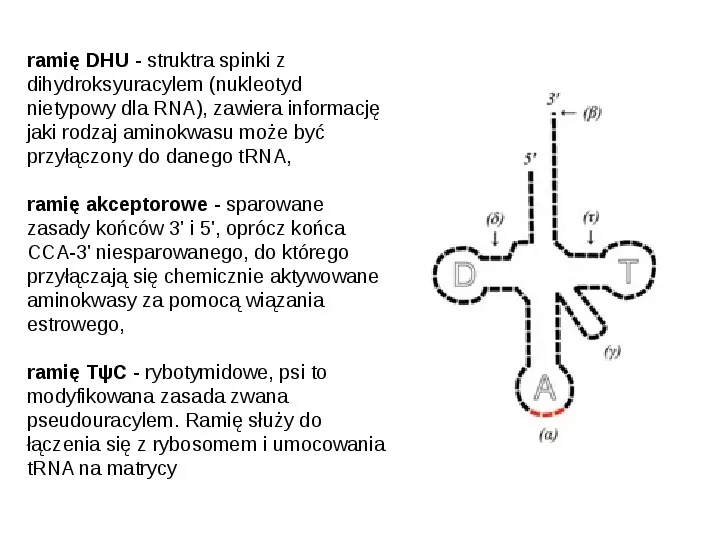
mniej więcej połowa nukleotydów tRNA jest sparowana i tworzy dwuniciowe helisy 4 lub 5 grup nieparujących się zasad: koniec CCA, 3 pętle: antykodonu, dihydrouracylu (zawiera kilka cząsteczek DHU) pętla TC oraz ramię dodatkowe o zmiennej liczbie nukleotydów. pętla antykodonu zawiera 7 zasad 10 25 nukleotydów w poszczególnych tRNA ulega modyfikacjom znanych jest ok. 80 różnych modyfikacji nukleotydów w tRNA
Slide 58
ramię DHU - struktra spinki z dihydroksyuracylem (nukleotyd nietypowy dla RNA), zawiera informację jaki rodzaj aminokwasu może być przyłączony do danego tRNA, ramię akceptorowe - sparowane zasady końców 3 i 5, oprócz końca CCA-3 niesparowanego, do którego przyłączają się chemicznie aktywowane aminokwasy za pomocą wiązania estrowego, ramię TψC - rybotymidowe, psi to modyfikowana zasada zwana pseudouracylem. Ramię służy do łączenia się z rybosomem i umocowania tRNA na matrycy
Slide 59
ramię zmienne - tylko w niektórych tRNA pętla antykodonowa - odpowiedzialna za rozpoznanie i związanie z kodonem w mRNA. Sekwencja antykodonowa rozpoznaje komplementarny tryplet nukleotydów tworzących kodon, na cząsteczce mRNA (w taki sposób następuje odczyt informacji genetycznej)
Slide 60
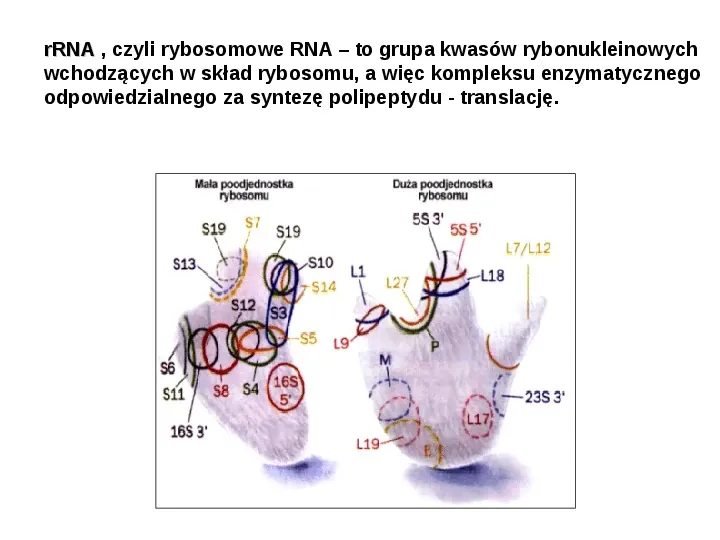
rRNA , czyli rybosomowe RNA to grupa kwasów rybonukleinowych wchodzących w skład rybosomu, a więc kompleksu enzymatycznego odpowiedzialnego za syntezę polipeptydu - translację.
Slide 61
Cząsteczki RNA mogą pełnić różne funkcje nośnik informacji składniki aparatu syntezy białka cząsteczki transportujące i aktywujące regulatory syntezy białka regulatory struktury chromatyny (chromosomów) regulatory syntezy RNA enzymy
Slide 62
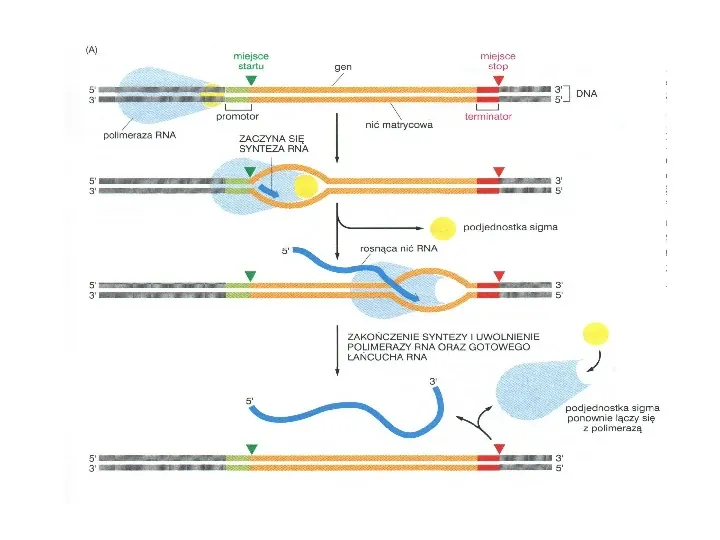
Transkrypcja pierwszy krok jaki podejmują komórki w celu odczytania instrukcji genetycznej polega na przepisaniu określonej części sekwencji DNA na sekwencję RNA
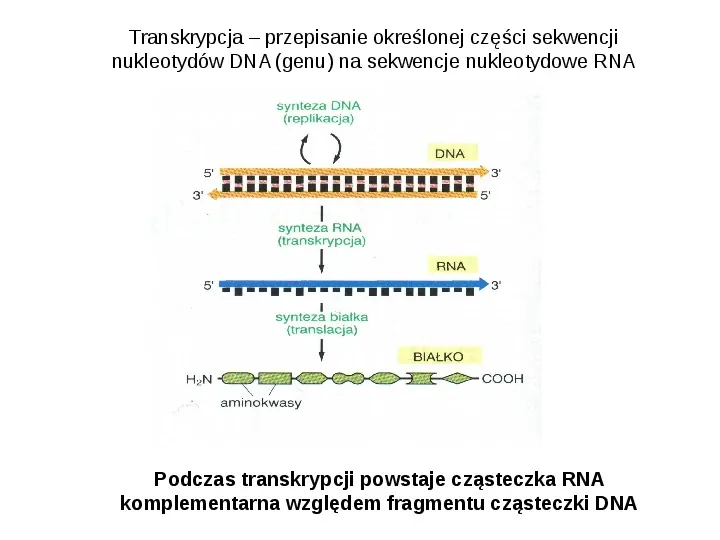
Slide 63
Transkrypcja przepisanie określonej części sekwencji nukleotydów DNA (genu) na sekwencje nukleotydowe RNA Podczas transkrypcji powstaje cząsteczka RNA komplementarna względem fragmentu cząsteczki DNA
Slide 64
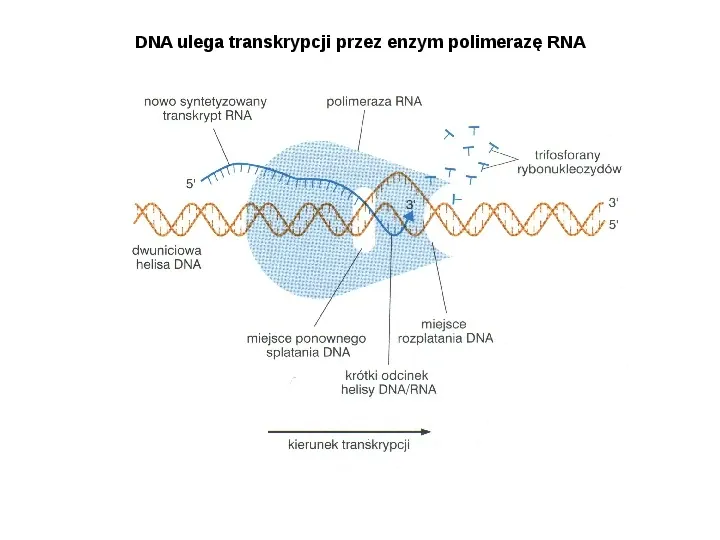
DNA ulega transkrypcji przez enzym polimerazę RNA
Slide 65
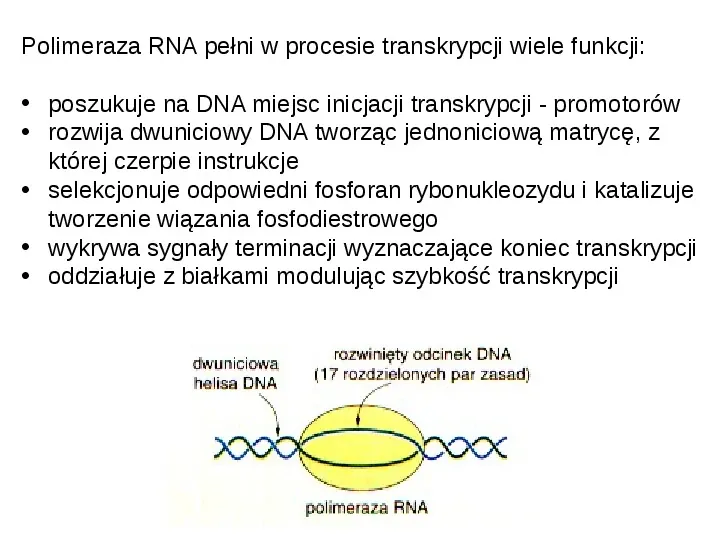
Polimeraza RNA pełni w procesie transkrypcji wiele funkcji: poszukuje na DNA miejsc inicjacji transkrypcji - promotorów rozwija dwuniciowy DNA tworząc jednoniciową matrycę, z której czerpie instrukcje selekcjonuje odpowiedni fosforan rybonukleozydu i katalizuje tworzenie wiązania fosfodiestrowego wykrywa sygnały terminacji wyznaczające koniec transkrypcji oddziałuje z białkami modulując szybkość transkrypcji
Slide 67
Pozostałe, ale nie mniej ważne, ustalenia dotyczące transkrypcji powstające cząsteczki RNA nie są gotowe do działania i muszą ulec obróbce (dojrzewaniu) transkrypcja prowadzi do powstania różnych typów cząsteczek RNA, pełniących różne funkcje w komórkach eukariotycznych różne typy cząsteczek RNA powstają dzięki działaniu różnych polimeraz RNA
Slide 68
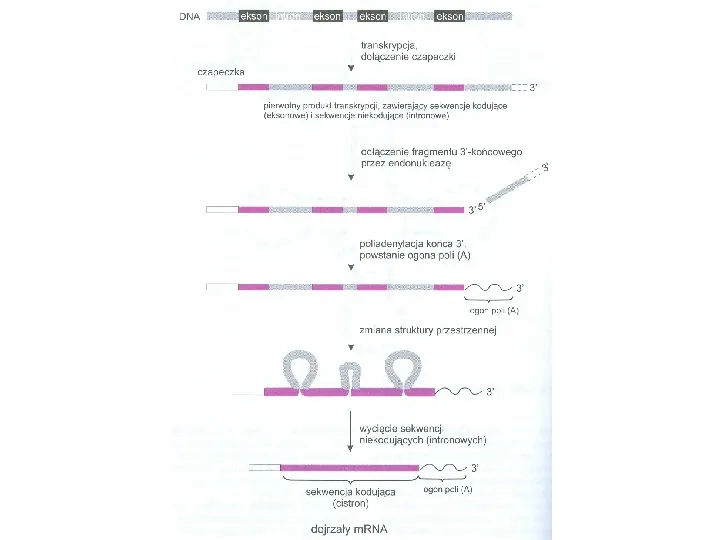
Przykłady sposobów obróbki pierwotnego transkryptu RNA modyfikacja końca 5 (synteza kapu) - przyłączenie guanozyny do nukleotydu znajdującego się na początku transkryptu i jego metylacji. Proces ten dotyczy niedojrzałej cząsteczki mRNA modyfikacja końców 3 polikondensacja, przyłączenie tzw. ogona poli(A). Niektóre transkrypty pierwotne zawierają po stronie końca 3 setki nukleotydów nieobecnych w dojrzałym mRNA. Są one odłączane przez nukleazę. Polimeraza poli A najpierw rozcina transkrypt w miejscu o specyficznej sekwencji nukleotydów a następnie dołącza do niego około 250 reszt AMP. W taki sposób tworzony jest ogon mający długość od 50 do 250 reszt adenylowych.
Slide 69
splicing splicing (mRNA) usunięcie wszystkich sekwencji intronowych niekodujących przez endonukleazy i połączenie eksonów w jedną całość
Slide 71
Translacja odkodowanie informacji zawartej w RNA i na tej podstawie utworzenie łańcucha polipeptydowego
Slide 72
Istotą procesu translacji jest tworzenie wiązań peptydowych pomiędzy kolejnymi konkretnymi aminokwasami zgodnie z informacją zapisaną kodem genetycznym w genie kodującym dany łańcuch polipeptydowy
Slide 73
TRANSLACJA BIOSYNTEZA BIAŁKA Translacja przepisanie informacji zawartej w mRNA na cząsteczki efektorowe powstanie łańcuchów polipeptydowych utworzenie pierwszorzędowej struktury białek Biosynteza białka ogół procesów prowadzących do powstania NATYWNEJ struktury białka. obejmuje: translację fałdowanie potranslacyjne modyfikacje
Slide 74
ETAPY TRANSLACJI: 1. INICJACJA obejmuje etapy przed utworzeniem pierwszego wiązania peptydowego. Obejmuje związanie mRNA do rybosomu oraz utworzenie kompleksu inicjującego. Jest to najwolniejszy etap translacji, decydujący o szybkości całej translacji i najsilniej regulowany. 2. ELONGACJA obejmuje reakcje od utworzenia pierwszego aż po ostatnie wiązanie peptydowe 3. TERMINACJA obejmuje uwolnienie polipeptydu z rybosomu oraz dysocjację rybosomów na podjednostki. Energia na różnych etapach translacji jest dostarczana przez hydrolizę GTP.
Slide 75
Co jest potrzebne do translacji? CO? PO CO? mRNA matryca tRNA funkcja adaptorowa; konieczne, bo aminokwasy nie rozpoznają kodonów funkcja adaptorowa; syntetazy aminoacylo-tRNA konieczne, bo aminokwasy nie rozpoznają kodonów rybosomy środowisko; enzym aminokwasy substraty czynniki białkowe regulatory
Slide 76
Rybosomy ogromne kompleksy enzymatyczne o złożonej strukturze prowadzące reakcję tworzenia wiązań peptydowych w oparciu o matrycę mRNA rRNA i białka rybosomów tworzą jedną interaktywną strukturę są miejscem grupującym składniki uczestniczące w biosyntezie białka struktury stanowiące środowisko kontrolujące oddziaływania pomiędzy mRNA i aminoacylo-tRNA rybosom posiada kilka miejsc aktywnych; każde z nich jest utworzone przez poszczególne grupy białek i odcinki rRNA.
Slide 77
Rybosom przesuwa się wzdłuż mRNA dokonując kodon po kodonie tłumaczenia sekwencji nukleotydowej na sekwencję aminokwasową
Slide 78
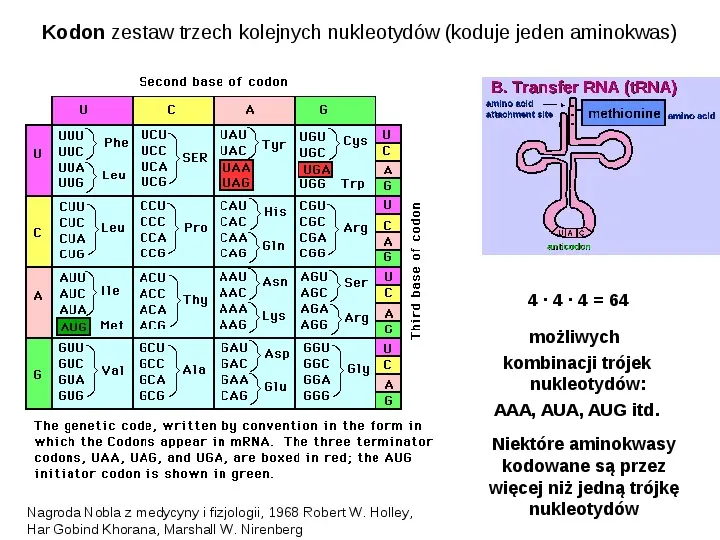
Kodon zestaw trzech kolejnych nukleotydów (koduje jeden aminokwas) 4 4 4 64 możliwych kombinacji trójek nukleotydów: AAA, AUA, AUG itd. Nagroda Nobla z medycyny i fizjologii, 1968 Robert W. Holley, Har Gobind Khorana, Marshall W. Nirenberg Niektóre aminokwasy kodowane są przez więcej niż jedną trójkę nukleotydów
Slide 79
Etapy odczytywania informacji zawartej w mRNA: 1. aminoacylacja tRNA przyłączenie aminokwasu do odpowiedniego tRNA dzięki syntetazom aminoacylo-tRNA. Dla każdego aminokwasu istnieje odrębna syntetaza 2. oddziaływania antykodon tRNA : kodon mRNA na rybosomie
Slide 80
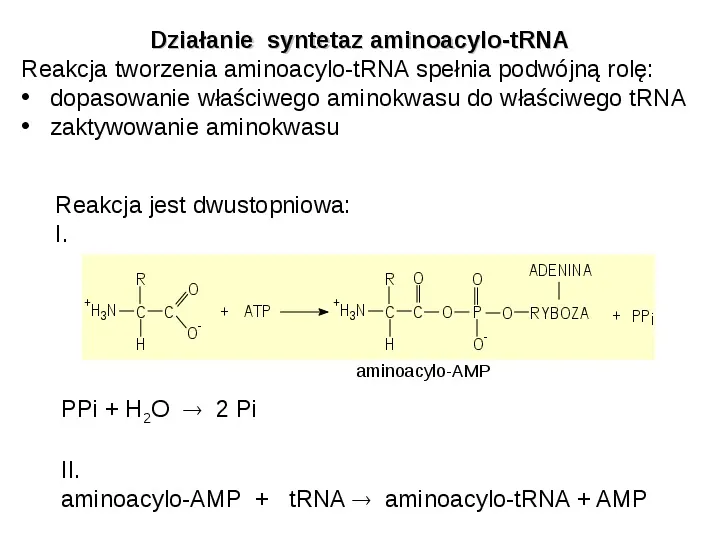
Działanie syntetaz aminoacylo-tRNA Reakcja tworzenia aminoacylo-tRNA spełnia podwójną rolę: dopasowanie właściwego aminokwasu do właściwego tRNA zaktywowanie aminokwasu Reakcja jest dwustopniowa: I. R H3 N C H O C - ATP O H3N R O C C H ADENINA O O P O RYBOZA PPi - O aminoacylo-AMP PPi H2O 2 Pi II. aminoacylo-AMP tRNA aminoacylo-tRNA AMP
Slide 81
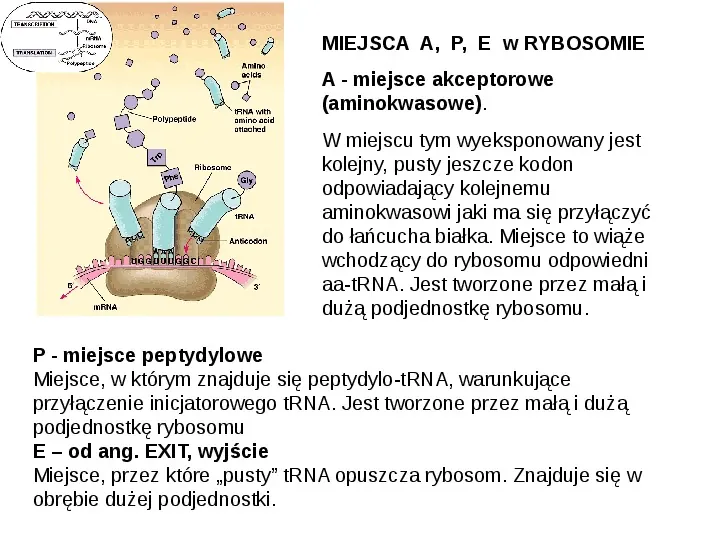
MIEJSCA A, P, E w RYBOSOMIE A - miejsce akceptorowe (aminokwasowe). W miejscu tym wyeksponowany jest kolejny, pusty jeszcze kodon odpowiadający kolejnemu aminokwasowi jaki ma się przyłączyć do łańcucha białka. Miejsce to wiąże wchodzący do rybosomu odpowiedni aa-tRNA. Jest tworzone przez małą i dużą podjednostkę rybosomu. P - miejsce peptydylowe Miejsce, w którym znajduje się peptydylo-tRNA, warunkujące przyłączenie inicjatorowego tRNA. Jest tworzone przez małą i dużą podjednostkę rybosomu E od ang. EXIT, wyjście Miejsce, przez które pusty tRNA opuszcza rybosom. Znajduje się w obrębie dużej podjednostki.
Slide 82
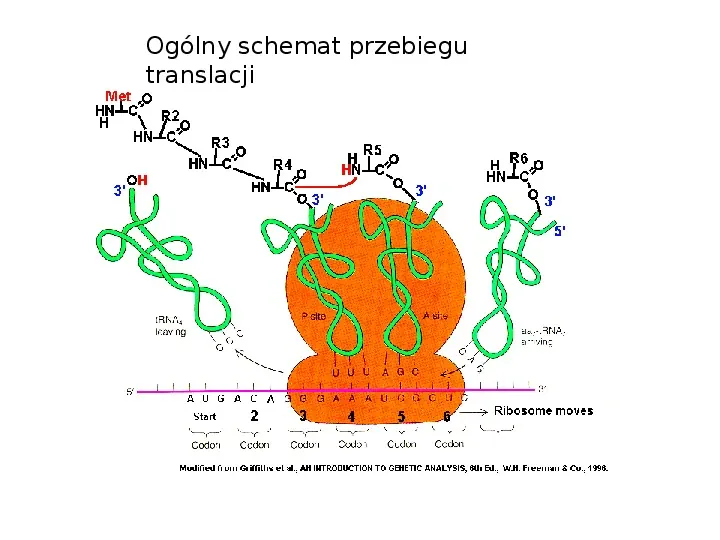
Ogólny schemat przebiegu translacji
Slide 83
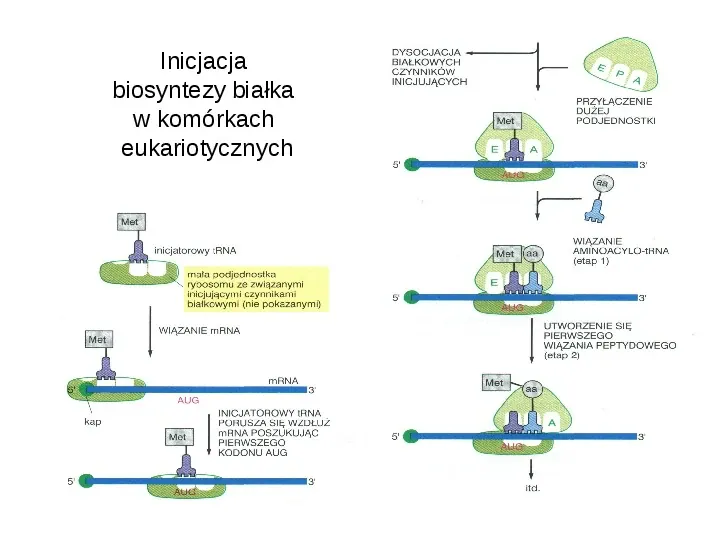
ETAPY INICJACJI TRANSLACJI mała podjednostka rybosomu wiąże inicjatorowy tRNA - ten, który niesie pierwszy aminokwas łańcucha polipeptydowego (metionina) mała podjednostka rybosomu jest rekrutowana do mRNA i umieszczana w miejscu kodonu inicjacji (initiation codon AUG) przyłącza się duża podjednostka tworzy się cały RYBOSOM
Slide 84
CZYNNIKI INICJUJĄCE (INITIATION FACTORS, IF) Są to białkowe czynniki uczestniczące tylko w inicjacji. Wiążą się tylko z małą podjednostką rybosomu i tylko do czasu jej asocjacji z dużą podjednostką. Wtedy oddysocjowują. NIE MA ICH W RYBOSOMIE 70S (80S) Ta aktywność odróżnia IF od strukturalnych (integralnych) białek rybosomów. Eukariotyczne czynniki inicjujące oznaczamy eIF (eukaryotic initiation factors)
Slide 85
ROZPOZNAWANIE KODONU INICJACYJNEGO U EUKARYOTA W 90 pierwszy AUG od 5 końca mRNA jest kodonem inicjacyjnym. Czasem jest to drugi AUG; czasem (wyjątkowo!) inny kodon: (CUG, ACG lub GUG). JAKI JEST MECHANIZM WYBORU PRZEZ RYBOSOM KODONU INICJACYJNEGO? nie zawsze wystarczy sama sekwencja AUG, aby zatrzymać skanowanie mRNA przez małą podjednostkę. Właściwa AUG jest odczytywana w kontekście innych (sąsiadujących z nią) nukleotydów. Optymalna sekwencja to: G C C A(G) C C AUG G Najważniejsze są: puryna o 3 nukleotydy przed AUG i nukleotyd guanylowy tuż za trójką AUG. Wtedy inicjacja translacji jest najwydajniejsza. Ta sekwencja to tzw. sekwencja Kozak
Slide 86
Inicjacja biosyntezy białka w komórkach eukariotycznych
Slide 87
INICJATOROWY tRNA Synteza białka zaczyna się od metioniny metionina jest pierwszym aminokwasem tworzącego się łańcucha polipeptydowego Sygnał stanowi inicjatorowy kodon AUG (u bakterii także GUG (8) (50 wydajności AUG) i UUG (1) (niska wydajność) Istnieją dwa typy tRNA mogących wiązać ten aminokwas i ten kodon (AUG): Uczestniczący w elongacji: Uczestniczący w inicjacji: Ta sama syntetaza aminoacylo-tRNA prowadzi obie reakcje (przyłącza resztę metioniny do obu rodzajów tRNA)
Slide 88
Dlaczego istnieją różne tRNA dla pierwszej metioniny i pozostałych reszt metioniny? fmet-tRNAf nie może przyłączyć się do miejsca A rybosomu. met-tRNAm nie może przyłączyć się do miejsca P rybosomu
Slide 89
ELONGACJA Rozpoczyna się od wprowadzenia do rybosomu drugiego aminoacylo-tRNA Kończy się z chwilą osiągnięcia przez rybosom kodonu STOP. Istota elongacji polega na tworzeniu wiązań peptydowych i translokacji rybosomu względem mRNA. Przebiega podobnie u Prokaryota i Eukaryota. Uczestniczą w niej cytoplazmatyczne czynniki elongacyjne: Prokaryota Eukaryota EF-Tu EF-Ts EF- G eEF-1A eEF-1B eEF-2
Slide 90
KONTROLA JAKOŚCI Konformacja miejsca A pasuje do każdego aa-tRNA do miejsca A wejść może każdy aa-tRNA. Pozostać powinien tylko taki, którego antykodon pasuje do kodonu. Jeśli wejdzie nieprawidłowy aa-tRNA to brak lub bardzo słabe oddziaływanie kodonantykodon powoduje, że nieprawidłowy aa-tRNA oddysocjowuje z miejsca A. Musi to nastąpić zanim zostanie utworzone wiązanie peptydowe. Podczas translacji nie zachodzi korekta (proof-reading) charakterystyczna dla procesu replikacji wbudowywanie aminokwasu musi być od razu prawidłowe. Fakt, że obecność EF-Tu uniemożliwia utworzenie wiązania peptydowego daje CZAS na to, aby nieprawidłowy aa-tRNA oddysocjował z miejsca A rybosomu. Błędnie wprowadzony aminokwas: 110 000.
Slide 91
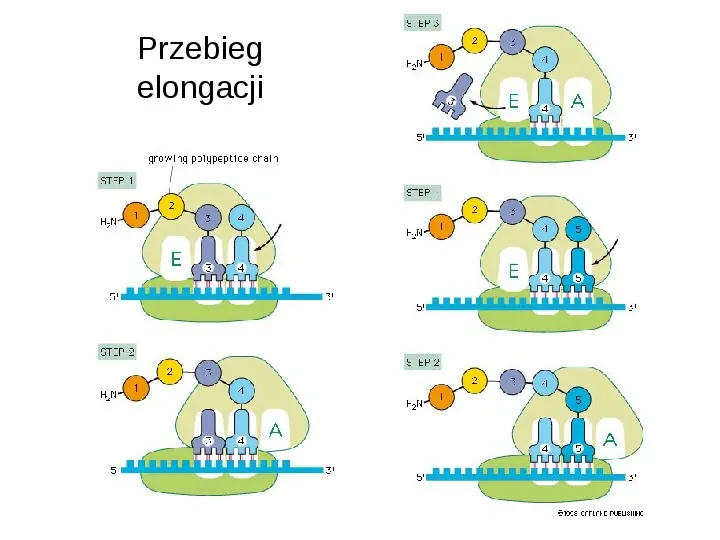
UTWORZENIE WIĄZANIA PEPTYDOWEGO Rosnący polipeptyd związany jest z tRNA znajdującym się w miejscu P na rybosomie. Z miejscem A wiąże się tRNA noszący następny aminokwas Karboksylowy koniec łańcucha polipeptydowego zostaje odłączony od tRNA w miejscu P i połączony z wolną grupą aminową reszty aminokwasu związanego z tRNA w miejscu A. Reakcja ta jest katalizowana przez peptydylotransferazę Reakcja jest sprzężona z przemieszczeniem się wolnego tRNA z miejsca P do E a peptydylo-tRNA miejsca A do P, mRNA przesuwa się o trzy nukleotydy
Slide 92
TRANSLOKACJA Następuje po utworzeniu wiązania peptydowego W wyniku translokacji deacylowany tRNA przenosi się do miejsca E. Peptydylo-tRNA przenosi się z miejsca A do miejsca P. Miejsce A zostaje wolne dla następnego aa-tRNA.
Slide 93
Przebieg elongacji
Slide 94
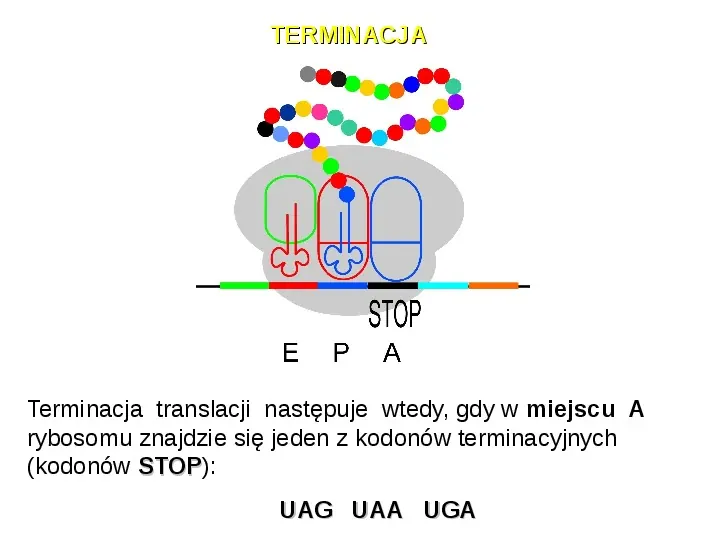
TERMINACJA Terminacja translacji następuje wtedy, gdy w miejscu A rybosomu znajdzie się jeden z kodonów terminacyjnych (kodonów STOP): STOP UAG UAA UGA
Slide 95
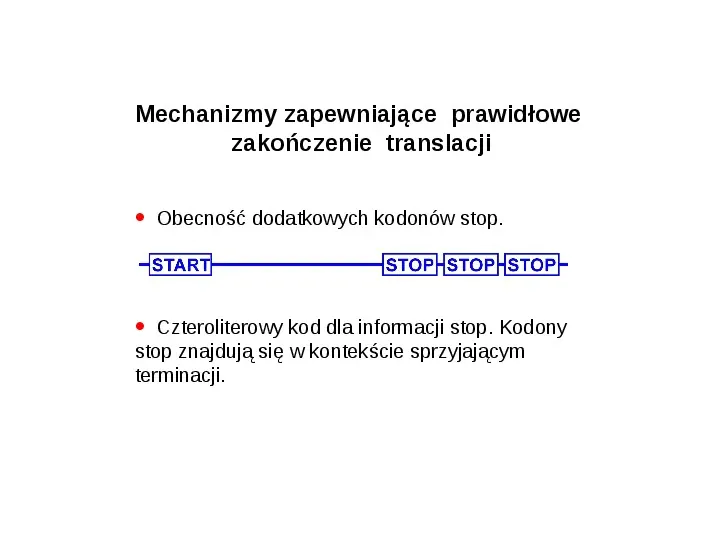
Mechanizmy zapewniające prawidłowe zakończenie translacji Obecność dodatkowych kodonów stop. Czteroliterowy kod dla informacji stop. Kodony stop znajdują się w kontekście sprzyjającym terminacji.
Slide 96
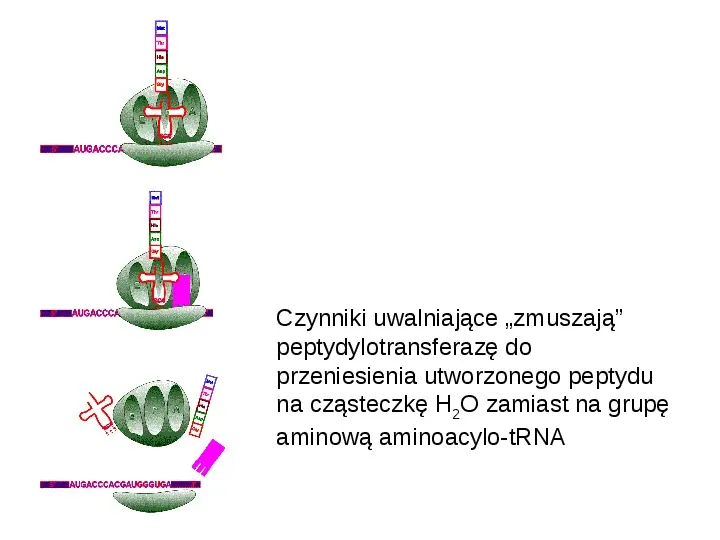
Czynniki uwalniające zmuszają peptydylotransferazę do przeniesienia utworzonego peptydu na cząsteczkę H2O zamiast na grupę aminową aminoacylo-tRNA
Slide 97
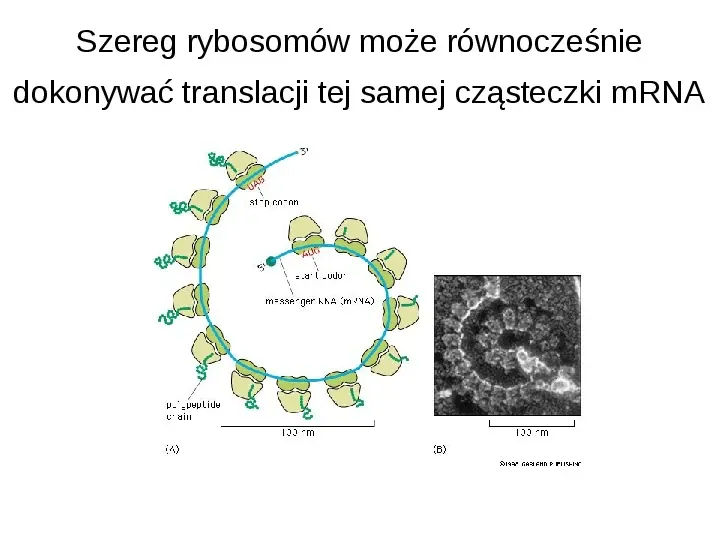
Szereg rybosomów może równocześnie dokonywać translacji tej samej cząsteczki mRNA
Dane:
- Liczba slajdów: 97
- Rozmiar: 10.29 MB
- Ilość pobrań: 5235
- Ilość wyświetleń: 34021